Новые способы работы с информацией
Новые способы работы с информацией
Одна из областей, в которых наши знания остаются до обидного неполными, — это изучение той роли, которую играют молекулы РНК в обработке генетической информации. Биологи то и дело открывают новые клеточные «информационные технологии», в которых РНК оказывается главным действующим лицом, и конца этим открытиям пока не видно.
—————
Новооткрытые функции РНК подтверждают теорию абиогенеза (самозарождения жизни). Почему раньше биологи не замечали множества разнообразных функций, выполняемых в клетке молекулами РНК? Может быть, слишком привыкли думать, что «всю работу в клетке делают белки»? Похоже на то. Как только ученые осознали, что жизнь началась с РНК (это понимание пришло, как мы помним, в середине 80-х годов XX века), стало ясно также и то, что теория РНК-мира имеет проверяемое следствие. Из нее следует, что, если хорошенько поискать, в современных живых клетках могут найтись ранее незамеченные «следы» эпохи РНК-мира — в том числе разные неожиданные функции, выполняемые молекулами РНК. Это следствие блестяще подтвердилось, и новые открытия продолжают его подтверждать по сей день, так что и конца не видно. Это одно из тех обстоятельств, которые все больше убеждают нас в том, что жизнь действительно возникла естественным путем из неживой материи. Почему? Судите сами.
Из теории естественного происхождения жизни (абиогенеза) следовало, что должна существовать молекула, с которой «все началось», — молекула, способная одновременно справляться и с «работой», и с хранением наследственной инофрмации. Это было проверямое следствие — единственный реальный способ проверить научную теорию, которую нельзя подтвердить или опровергнуть прямым наблюдением (а такова большая часть научных теорий). Проверяемое следствие — чрезвычайно ценная вещь! Именно по наличию или отсутствию проверяемых следствий всегда можно отличить научную теорию от ненаучной. Например, креационизм не является научной теорией как раз потому, что не имеет проверяемых следствий. Ведь Бог мог сотворить жизнь и Вселенную с абсолютно любым строением и свойствами. Какое бы неожиданное свойство мы ни обнаружили, всегда можно сказать: это так, потому что так было угодно Богу. Мотивы высшего разума мы постичь не можем, поэтому не можем и предсказать ничего конкретного о тех областях реальности, которые еще не изучены. Наука избегает таких теорий, из которых ничего конкретного не следует. Из теории абиогенеза, напротив, следовало нечто вполне конкретное: должна существовать молекула с такими-то свойствами. Подходящую молекулу нашли — ею оказалась РНК. Проверяемое следствие подтвердилось, и теория абиогенеза заработала себе очередной большой и жирный «плюсик». Теория РНК-мира после этого стала важной составной частью теории абиогенеза. Из нее, в свою очередь, вытекали новые проверяемые следствия, которые сегодня подтверждаются, и тем самым новые «плюсики» зарабатывает и теория РНК-мира, и вмещающая ее теория абиогенеза.
—————
Взять, к примеру, тот же альтернативный сплайсинг. Каким образом клетка «решает», какой из вариантов сплайсинга нужно выбрать в данной ситуации (и, следовательно, какой из вариантов белка синтезировать)? Об этом пока известно очень мало. Удалось выяснить, что такая регуляция требует участия особых белков — регуляторов сплайсинга. Не было оснований думать, что сплайсинг регулируется чем-то еще, кроме белков. И вдруг ученые из Йельского университета (США) публикуют статью[108], в которой описан совершенно иной способ регуляции, где белки не участвуют вообще. Ключевую роль в нем играет сама молекула РНК, подвергающаяся сплайсингу.
Способность молекулы РНК самостоятельно определять свою судьбу и выбирать способ, каким она будет перекроена, определяется наличием в одном из ее некодирующих участков (интронов) специфической последовательности нуклеотидов, которая сама собой сворачивается в особую трехмерную структуру — РНК-переключатель. О том, что это такое, мы уже рассказывали в главе «Происхождение жизни».
Исследование проводилось на грибе Neurospora crassa, известном широкой публике как розовая хлебная плесень. По иронии судьбы, на этом же объекте в 40-е годы прошлого века были получены сенсационные результаты, позволившие сформулировать принцип «один ген — один белок». Сейчас на нейроспоре изучают альтернативный сплайсинг — явление, опровергающее (или, лучше сказать, уточняющее и расширяющее) этот замечательный принцип.
У нейроспоры, как и у ряда других эукариот, в генах, участвующих в биосинтезе тиамина (витамина B1), были обнаружены участки, сходные с известными бактериальными РНК-переключателями, которые реагируют на производное тиамина — тиамин-пирофосфат. Большинство известных РНК-переключателей действуют по принципу отрицательной обратной связи. Они реагируют на вещество, синтезируемое белковым продуктом данного гена, и при достаточно высокой концентрации этого вещества отключают ген.
Примерно то же самое наблюдалось и в данном случае. Повышение концентрации тиамин-пирофосфата в клетках гриба приводит к снижению производства белков, ответственных за синтез тиамина. Было показано, что если удалить из соответствующих генов участки, похожие на бактериальные РНК-переключатели, то производство тиамин-синтезирующих белков перестает зависеть от концентрации тиамин-пирофосфата.
Таким образом, стало ясно, что участки грибных генов, похожие на РНК-переключатели, действительно являются таковыми. Оставалось лишь выяснить механизм их действия, то есть понять, как они блокируют работу «своих» генов. У бактерий РНК-переключатели делают это либо на этапе транскрипции (первичного «прочтения» гена), либо на этапе трансляции — синтеза белка на матрице мРНК.
У эукариот, как выяснилось, дело обстоит иначе — работа гена блокируется на этапе сплайсинга. Бактериям это недоступно, поскольку у бактерий сплайсинга нет (почти нет, если быть точным). Тиаминовый РНК-переключатель в генах Neurospora crassa располагается в первом интроне, недалеко от начала гена. Если в клетке мало тиамин-пирофосфата, РНК-переключатель «приклеивается» одной из своих петель к строго определенному месту на молекуле мРНК. Это место является одним из потенциальных мест сплайсинга, то есть именно здесь в ходе сплайсинга молекула мРНК может быть разрезана. Однако приклеившийся РНК-переключатель не позволяет этого сделать, и молекула разрезается в другом подходящем месте по соседству. В результате формируется «правильная» зрелая мРНК, на основе которой синтезируется полноценный белок.
Если же в клетке много тиамин-пирофосфата, это вещество присоединяется к РНК-переключателю и изменяет его форму. Переключатель «отклеивается» от места сплайсинга и перестает его защищать. Тогда молекула РНК режется именно в этом месте, которое раньше прикрывалось РНК-переключателем. Это в конечном счете приводит к формированию «бракованной» зрелой мРНК, на базе которой полноценный белок синтезировать невозможно.
Таким образом, РНК-переключатель в зависимости от концентрации тиамин-пирофосфата направляет сплайсинг по одному из двух альтернативных путей.
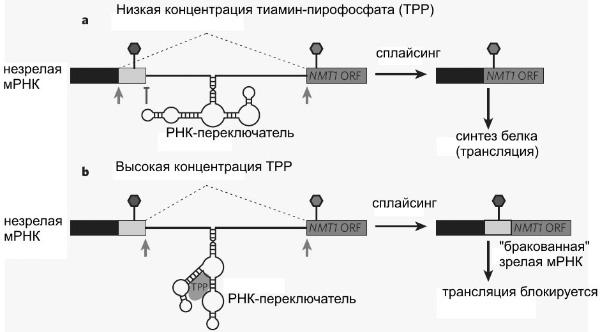
На этом рисунке показано, как РНК-переключатель регулирует альтернативный сплайсинг у розовой хлебной плесени (на примере гена NMT1). Участок мРНК, вырезаемый при сплайсинге, отмечен пунктирными линиями и серыми стрелками. При низкой концентрации тиамин-пирофосфата РНК-переключатель «защищает» потенциальный сайт (место) сплайсинга, отмеченный значком в виде буквы «Т». В результате при сплайсинге вместо этого сайта используется другой, расположенный по соседству (серая стрелка). Участок мРНК, отмеченный белым цветом, не попадает в зрелую мРНК. При высокой концентрации ТРР это вещество связывается с РНК-переключателем и меняет его конфигурацию. В результате молекула РНК режется в том месте, которое раньше было прикрыто РНК-переключателем, белый участок попадает в зрелую РНК и «портит» ее.
Судя по некоторым косвенным признакам, регуляция сплайсинга при помощи РНК-переключателей может быть довольно широко распространена у эукариот. Чтобы проверить это предположение, необходима разработка эффективных методов поиска РНК-переключателей в эукариотических геномах — эти методы пока еще далеки от совершенства.
Еще одна неожиданная функция РНК обнаружилась недавно в ходе изучения механизмов репарации — починки повреждений в ДНК. Оказалось, что молекулы РНК могут играть роль матриц, информация с которых переписывается в поврежденную молекулу ДНК взамен утерянной[109]. Процесс основан на обратной транскрипции (как мы помним из предыдущей главы, так называют переписывание информации из РНК в ДНК, то есть синтез ДНК на РНК-матрице). Изобретение обратной транскрипции, между прочим, должно было стать важным переломным моментом в развитии РНК-мира, поскольку позволило РНК-организмам перейти к хранению наследственной информации в более надежной и стабильной форме молекул ДНК. В предыдущих главах мы упоминали несколько случаев использования обратной транскрипции современными организмами: это перемещение и размножение ретротранспозонов и ретровирусов, образование ретропсевдогенов, восстановление укорачивающихся при каждом клеточном делении кончиков хромосом — теломер. И вот еще один пункт добавился к этому списку — репарация ДНК.
Если молекула ДНК повреждена — например, подверглась разрыву, — для ее починки необходима матрица, в которой последовательность нуклеотидов соответствует исходному, «правильному» состоянию поврежденного участка. Ранее считалось, что в качестве таких матриц всегда используются другие молекулы ДНК.
Потом выяснилось, что иногда эти ДНК-матрицы синтезируются путем обратной транскрипции на основе РНК при участии ретротранспозонов.
Наконец, совсем недавно ученые из Национального института экологии здоровья (США) сумели показать, что репарация возможна и непосредственно на основе РНК-матриц без предварительного изготовления ДНК-матрицы и без участия специфических ферментов — обратных транскриптаз, кодируемых ретротранспозонами.
Исследователи искусственно вызывали у дрожжей разрыв хромосомы в одном и том же строго определенном месте. Затем в клетки добавляли искусственно синтезированные короткие молекулы РНК, последовательности нуклеотидов в которых соответствовали участкам поврежденной хромосомы по краям разрыва. Оказалось, что эта процедура повышает вероятность успешной «починки» разорванной хромосомы в 500 раз.
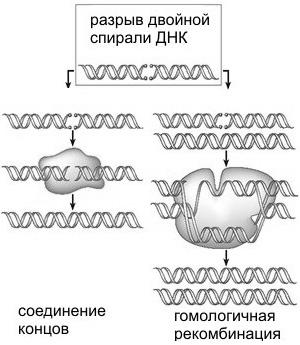
Два основных способа «починки» разрывов двойной спирали ДНК. Первый способ (негомологичное соединение концов) чреват неточностями — потерей или вставкой лишних нуклеотидов в районе разрыва. Второй более точен, но требует наличия «запасной копии» поврежденного фрагмента ДНК. Как выяснилось, эта запасная копия не обязательно должна быть двухцепочечной ДНК (как показано на рисунке), годится и одноцепочечная ДНК и даже РНК.
Если в середину молекулы РНК, служащей матрицей для репарации, ввести несколько лишних нуклеотидов, они потом обнаруживаются в «починенной» хромосоме как раз между сшитыми краями разрыва. Это свидетельствует о синтезе ДНК на матрице РНК, то есть об обратной транскрипции.
Кроме того, это говорит об отсутствии принципиальных преград для переписывания информации из РНК в ДНК в живых клетках, что может иметь большое значение для эволюции. Уже целый ряд фактов указывает на то, что молекулы РНК иногда могут служить чем-то вроде «резервных копий» особо важных «файлов», записанных в геномной ДНК, и при потере каких-то фрагментов информации (даже целых генов!) эти резервные копии могут идти в ход. Мы уже знаем три таких примера (описанные в главе «Управляемые мутации» геномные перестройки у инфузорий, парамутации, участие РНК в репарации), а есть и другие, не поместившиеся в эту книгу. Ясно, что все это открывает большие возможности для эволюции, хотя о том, в какой мере и для чего именно они используются, мы пока можем только гадать.
—————
Сравнение геномов человека и мыши помогло обнаружить новый способ регуляции работы генов. Ну и напоследок — еще одно недавнее открытие, которое как нельзя лучше показывает, что наши знания об устройстве и функционировании живой клетки до сих пор крайне неполны, а сложность и запутанность механизмов внутриклеточной регуляции далеко превосходит все, что мы еще недавно могли себе представить.
В основе нового способа генной регуляции, только что открытого биологами из Калифорнийского университета в Беркли, лежит уже знакомый нам альтернативный сплайсинг, а также еще один механизм, называемый nonsense-mediated mRNA decay (NMD). Это можно перевести как «разрушение мРНК, опосредуемое бессмыслицей». Данный механизм представляет собой нечто вроде «внутриклеточной цензуры». Он служит для уничтожения заведомо бессмысленных молекул РНК. Особые молекулярные системы, о которых пока мало что известно, идентифицируют зрелую (то есть прошедшую сплайсинг) мРНК как бессмысленную и приговаривают ее к уничтожению в том случае, если в ней имеется «преждевременный» стоп-кодон (три нуклеотида, сигнализирующие об окончании синтеза белка). В норме стоп-кодон должен располагаться в конце каждого гена. Но в результате мутации он может образоваться в середине гена. В этом случае синтез белка обрывается преждевременно, и весь ген становится бессмысленным. Именно для выявления и «обезвреживания» таких мутаций и существует система NMD.
Открытие, о котором идет речь, было сделано в ходе анализа так называемых «ультраконсервативных» участков генома. Как известно, геномы человека и мыши довольно сильно различаются. Однако имеются и довольно длинные (до многих сотен нуклеотидов) абсолютно идентичные участки. Ясно, что такая консервативность нуклеотидных последовательностей должна иметь какой-то смысл. По-видимому, она означает, что любое изменение этих участков неизменно оказывается вредным и для человека, и для мыши, поэтому естественный отбор быстро отсеивает таких мутантов.
Некоторые ультраконсервативные участки находятся между генами и могут выполнять регуляторные функции. Другие лежат прямо внутри генов, задевая как интроны, так и экзоны. Это уже более странно, поскольку обычно структура генов и белков довольно пластична, а многие нуклеотидные замены в пределах экзонов вообще не влияют на структуру кодируемого белка. Такие замены в ходе эволюции обычно накапливаются вполне свободно.
Ученые заметили, что ультраконсервативные участки имеются во всех без исключения генах семейства SR (splicing regulators). Эти гены кодируют белки, участвующие в регуляции альтернативного сплайсинга.
Детальный анализ показал, что гены SR сами подвержены альтернативному сплайсингу, причем в результате могут получаться такие мРНК, которые должны уничтожаться системой NMD. Оказалось, что у каждого SR-гена имеются два альтернативных сплайс-варианта. Один из них представляет собой «нормальную» мРНК, которая может служить матрицей для синтеза полноценного SR-белка. Второй содержит преждевременный стоп-кодон и должен уничтожаться цензурой. Эксперименты показали, что этот второй сплайс-вариант действительно активно уничтожается системой NMD.
Обнаруженный регуляторный контур работает по принципу отрицательной обратной связи. SR-белки могут контролировать альтернативный сплайсинг не только «чужих» мРНК, но и своих собственных. Чем больше производит клетка SR-белков, тем чаще они направляют сплайсинг своих мРНК по альтернативному пути, тем самым снижая темп производства новых SR-белков. Так поддерживается некоторое постоянство концентрации SR-белков в клетке.
Как мог возникнуть такой причудливый регуляторный контур? Ведь производство SR-белков вполне могло бы контролироваться каким-нибудь менее экзотическим способом — например, при помощи транскрипционных факторов. Видимо, в данном случае при создании контура обратной связи отбор просто «зацепился» за то, что ближе лежало, а именно за способность SR-белков контролировать альтернативный сплайсинг. Аналогичным образом, активность генов, кодирующих транскрипционные факторы, обычно регулируется не чем-нибудь, а транскрипционными факторами (хотя и исключений немало). Это характерный «почерк» эволюции, совсем не похожий на разумное проектирование, а похожий скорее на самосборку чего получится из чего попало[110].
Десять генов, составляющих SR-семейство, родственны друг другу. Общий предок людей и мышей уже имел все десять, но у него самого когда-то был предок только с одним исходным SR-геном, от которого произошли все остальные путем последовательных дупликаций. Самое удивительное, что механизм саморегуляции с участием альтернативного сплайсинга и цензуры, судя по всему, возникал у разных представителей SR-семейства параллельно и независимо. Это видно из того, что преждевременные стоп-кодоны образуются у них в ходе альтернативного сплайсинга разными способами. У одних SR-генов имеется особый экзон со стоп-кодоном, который в норме вырезается из мРНК вместе с интронами. У других к концу мРНК после «законного» стоп-кодона может быть пришита пара-тройка лишних экзонов, в результате чего законный стоп-кодон с точки зрения системы цензуры начинает выглядеть как преждевременный. Очевидно, SR-гены имеют высокую предрасположенность к формированию таких регуляторных контуров.
Ультраконсервативными у людей и мышей обычно являются те участки SR-генов, которые непосредственно прилегают к преждевременному стоп-кодону, — но не всегда и не только. Скорее всего, уникальный консерватизм этих участков обусловлен тем, что они необходимы для правильного сплайсинга. Проверить это предположение трудно, поскольку о молекулярных механизмах сплайсинга нам известно пока очень мало. Вместе с тем у асцидии Ciona (далекого родственника позвоночных животных) тоже есть SR-гены с альтернативными экзонами, несущими преждевременные стоп-кодоны. Нуклеотидные последовательности этих генов похожи на человеческие и мышиные, но все же не идентичны им. Это значит, что ультраконсервативность в данном случае не является абсолютной. Ситуация еще сильнее запуталась совсем недавно, когда удалось экспериментально показать, что удаление некоторых некодирующих ультраконсервативных участков из генома мышей совершенно не вредит их здоровью.
Не исключено, что новооткрытый способ генной регуляции распространен довольно широко и затрагивает отнюдь не только гены — регуляторы сплайсинга. Дальнейшие исследования должны пролить свет на этот вопрос.
На мой взгляд, данное открытие говорит прежде всего о том, как мало мы еще знаем о жизни клетки и о возможных путях ее эволюционных изменений. Сколько еще подобных открытий предстоит сделать, прежде чем у нас появится достаточно оснований для построения всеобъемлющей эволюционной теории, или, как сейчас говорят, «нового эволюционного синтеза»? Думаю, много.
(Источник: Liana F. Lareau, Maki Inada, Richard E. Green, Jordan C. Wengrod, Steven E. Brenner. Unproductive splicing of SR genes associated with highly conserved and ultraconserved DNA elements // Nature. 2007. V. 446. P. 926–929.)
—————
Более 800 000 книг и аудиокниг! 📚
Получи 2 месяца Литрес Подписки в подарок и наслаждайся неограниченным чтением
ПОЛУЧИТЬ ПОДАРОКДанный текст является ознакомительным фрагментом.
Читайте также
3. Способы роста ооцитов
3. Способы роста ооцитов Когда гоноциты попадают в половые железы, их не всегда можно отличить от соматических клеток — клеток стенки железы, между которыми они встраиваются. Но вот после ряда делений будущие женские половые клетки вступают на путь образования яйца. С
Глава 14 Homo erectus. Развитие мозга. Зарождение речи. Интонации. Речевые центры. Глупость и ум. Смех—плач, их происхождение. Обмен информацией в группе.
Глава 14 Homo erectus. Развитие мозга. Зарождение речи. Интонации. Речевые центры. Глупость и ум. Смех—плач, их происхождение. Обмен информацией в группе. Homo erectus оказался весьма пластичным «прачеловеком»: за миллион с лишним лет своего существования он все время
СПОСОБЫ ОБЩЕНИЯ
СПОСОБЫ ОБЩЕНИЯ Так муравьи, столкнувшись где-нибудь. Потрутся рыльцами, чтобы дознаться, Быть может, про добычу и про путь... А. Данте. Божественная комедия В прошлом мало у кого возникали сомнения в том, умеют ли муравьи или пчелы разговаривать друг с другом.
Бактерии тоже обмениваются генетической информацией
Бактерии тоже обмениваются генетической информацией Как я уже говорил в главе 2, инфузории, будучи всего лишь «простейшими» одноклеточными организмами, оказываются тем не менее сравнительно высокоразвитыми созданиями. В эстафете жизни они намного опередили многих
Способы расселения.
Способы расселения. Этот вопрос уже разбирался сэром Ч. Лайеллем и другими авторами. Я, со своей стороны, могу представить здесь очень краткий обзор наиболее важных фактов. Перемены в климате должны были оказать сильнейшее влияние на миграцию. Регион, в настоящее время
Глава 5 Новые гены, новые признаки
Глава 5 Новые гены, новые признаки В предыдущих главах мы уже познакомились с несколькими примерами появления эволюционных новшеств. Внимательный читатель заметил, что новые гены и новые признаки могут возникать разными способами. Пожалуй, нам пора эти способы
2.2. Стандартные способы получения воспроизводимой конфигурации ССП
2.2. Стандартные способы получения воспроизводимой конфигурации ССП Методические требования к регистрации ЭЭГ (установка электродов, выбор системы отведения, полосы пропускания усилителей, способы устранения артефактов) описаны в главе 2. Заметим, что для регистрации
Пастер создает новые методы и новые вакцины
Пастер создает новые методы и новые вакцины Бешенство — роковое заболевание, от которого не было спасения. Каждому человеку, укушенному бешеным животным, грозила неминуемая смерть. До получения Пастером вакцины против бешенства из каждых 100 человек, укушенных и
Новые методы, новые вакцины
Новые методы, новые вакцины Поиски методов получения новых живых вакцин проводятся на основе современных научных данных об изменчивости микроорганизмов. Раскрывая тончайшее строение и химический состав микробной клетки и вирусной частички, изучая их физиологию и
Способы вызывания гипноза
Способы вызывания гипноза Обращаясь к вопросу о вызывании гипноза, необходимо иметь в виду, что для этого с пользою могут служить различные способы, из которых одни могут считаться физиологическими, так как они действуют непосредственно на те или другие из
СПОСОБЫ ДРЕССИРОВКИ
СПОСОБЫ ДРЕССИРОВКИ Под способом понимают такое воздействие на животное, которое приводит к воспроизведению (появлению) нужных дрессировщику действий или сохранению необходимых поз.Хотя, по словам К. Прайор, «способов дрессировки существует столько, сколько
Способы рентгенологических исследований
Способы рентгенологических исследований а) Просвечивание (рентгеноскопия). Рентгеновские лучи в ветеринарной практике применяют для изучения и распознавания разных болезней у сельскохозяйственных животных. Этот метод исследования больных животных является
Способы размножения
Способы размножения Размножение — важнейший биологический процесс, обеспечивающий поддержание и увеличение численности вида, возможность его расселения и, в конечном итоге, успех борьбы за существование. В животном мире существует целый ряд способов размножения,
Разные способы защиты
Разные способы защиты Способы защиты насекомых от врагов бесконечны в своем многообразии. Тот, кто часто бывает на природе, мот заметить, что как только какое-либо животное становится многочисленным — будь то ящерица, птица, зверь, насекомое, оно сразу же делается менее
Различные способы создания неконтролируемой ситуации
Различные способы создания неконтролируемой ситуации Чтобы стрессорная ситуация оказалась неконтролируемой, воздействие должно отвечать одному или нескольким условиям, о которых мы говорили выше.Наименее интересным является случай со стимулом, к которому невозможно
Электрохимия и сигналы с информацией
Электрохимия и сигналы с информацией Итак, выше мы философски оценили наше тело — чтобы наше тело функционировало, требуется сила извне тела, поскольку тех сил, которые известны химии, для жизни тела явно недостаточно.И поскольку это явно не механические силы и, как